Hace poco menos de un año (el 11 de marzo para ser preciso), el Director General de la Organización Mundial de la Salud (OMS), Tedros Adhanom, declaró que la COVID-19 podría comenzar a calificarse como una pandemia.
Ante esto , el volumen de búsqueda relativo (VBR) relacionado al nuevo coronavirus se disparó y el consumo de información referente al nuevo virus se volvió masiva. Lamentablemente, y al mismo estilo que nos advirtió Huxley, nos atiborramos de irrelevancia y desinformación. El pseudoconocimiento comenzó a llegar sin mayores filtros y la propagación de datos mal interpretados, paparruchas e inferencias fantásticas se enquistaron en el imaginario popular bajo el amparo de figuras que proyectaban seguridad en su momento.
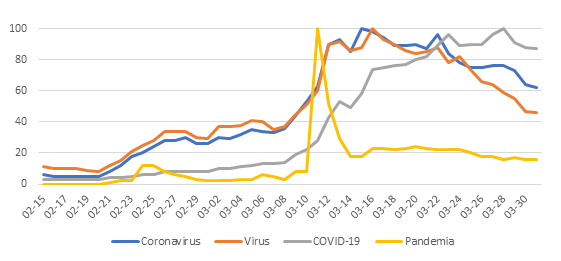
A nivel comunitario, era previsible que la incertidumbre e incongruencia de datos generara miedo, frustración y dañara nuestra percepción de autoeficacia para afrontar la situación a la que nos estábamos adentrando. Por tanto, no es extraño que el sector salud y la academia se enfrente ahora a una serie de mitos e imprecisiones referentes al SARS-CoV-2 y las acciones protectivas para evitar su contagio.
Table of Contents
El COVID-19 en el ámbito de la medicina veterinaria
Un análisis de la tendencia temporal para la búsqueda de información en salud nos indica que existe cierta preocupación en la población por la capacidad de los animales domésticos para contraer y contagiar la enfermedad (Figura 2).
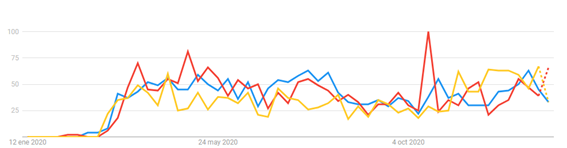
Figura 2.
Figura 2. Interés a lo largo del tiempo sobre el COVID y mascotas. (Rojo: Gato-COVID, Amarillo: hurón-COVD, Azul: perro-COVID) Fuente: Google trends.
Además, entre otras consultas, las medidas de control referentes al trato con animales fueron cuestionamientos comunes durante el estado de emergencia.
Por esto, y para que puedas basar en ciencia tus recomendaciones a estas y otras consultas sobre el tema, hemos preparado una serie de artículos en donde podrás encontrar todo lo que debes saber acerca del SARS-CoV-2, la COVID-19 y sus implicancias sobre el sector veterinario, así como un repaso de las enfermedades filogenéticamente cercanas que afectan a los animales.
Sin mayores preámbulos e inspirados en “El Arte de la guerra de Sun Tzu” como esquema de gestión de la información, iniciamos el proceso para comprender mejor esta enfermedad y al virus que la causa.
Diferencias entre el COVID-19 Y SARS-CoV-2
Aunque probablemente ya lo sepan, es importante tenerlo claro:
- COVID-19: la enfermedad causada por el virus (Coronavirus Viral Disease–2019)
- SARS-CoV-2: el agente etiológico de la enfermedad.
En otras palabras: La presencia de este virus en una persona u animal se denomina infección por SARS-CoV-2, mientras que el desarrollo de enfermedad a causa del virus (independientemente de la sintomatología o gravedad) se llama COVID-19.
¿Por qué es importante esta aclaración?
La presencia de un virus en cualquier organismo no siempre tiene implicancias clínicas. El desarrollo de una enfermedad depende del grado de afección que pueda generar el virus sobre el estado fisiológico del hospedero.
A veces la replicación de un virus en un hospedero no resulta en lesiones histológicas graves o relevantes, en el caso del SARS-CoV-2, aún no se terminan de entender los motivos de una infección sin síntomas.
El asunto aún se encuentra en el terreno de la especulación pero se propone que la severidad de los cuadros podría ser dosis dependiente y que la adaptación del virus a los receptores ACE2h aún no es completa (las nuevas cepas de las que estamos teniendo conocimiento mutaron justamente en los dominios receptor-obligatorio).
Orígenes del SARS-CoV-2
El SARS-CoV-2, inicialmente llamado 2019-nCoV fue descubierto por el grupo de investigación del Dr. Zhu en muestras de pacientes hospitalizados en Wuhan entre diciembre del 2019 y enero del 2020. Aunque esto sea ignorado por algunos medios y comunicadores, el virus sí fue aislado antes de que inicie la pandemia. De hecho, se utilizaron 3 líneas celulares para este fin: células epiteliales de vías respiratorias humanas, las Vero E6 y Huh-7.
Todos los cultivos celulares en ese primer estudio evidenciaron efectos citopáticos con la diferencia de que las células del epitelio respiratorio solo demoraron 92 horas mientras que el resto 6 días desde su inoculación. Estos cultivos fueron los que hicieron posible la recolección de viriones para su caracterización estructural por microscopía electrónica de transmisión.
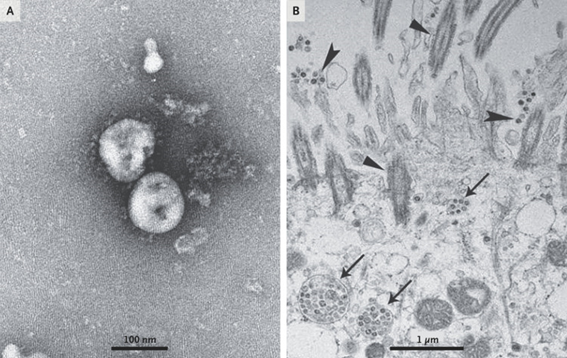
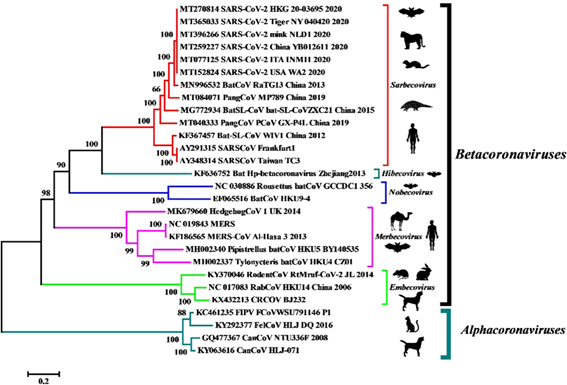
La identificación molecular del virus se dio mediante el estudio metagenómico del ARN de muestras del lavado bronqueo alveolar de los pacientes y analizó los más de 10 millones de reads obtenidos desde las plataformas de illuminia y nanopore de las que luego se logró reconstruir un genoma completo de 29891 pares de bases con 79.6% de identidad con el SARS-CoV BJ01. Un análisis bioinformático posterior reveló que el ancestro común más cercano del que tendríamos conocimiento es el RaTG13, un beta-coronavirus descubierto en heces de murciélagos en el 2013.
Aunque sólo mantenga 1 de los 6 residuos importantes para el dominio de unión al receptor del gen S, el RaTG13 también usa como entrada celular a la enzima convertidora de angiotensina 2 (ACE-2) y guarda una identidad del 90.55% con el segundo pariente más cercano, el coronavirus del pangolín (Pangolin-CoV MP789).
Este otro coronavirus, a pesar de tener sólo un 91.02% de identidad a nivel de genoma completo con el SARS-CoV-2, exhibe una consistencia perfecta con los 6 residuos que mantienen contacto físico durante la interacción con los receptores ACE-2 humanos, dándonos la idea de que estas dos especies podrían estar involucradas en el origen evolutivo de la pandemia.
Sobre el origen antropogénico del SARS-CoV-2 como accidente de laboratorio o bajo la manipulación genética creemos que, aunque sea poco probable, la discusión debe continuar para esclarecer cualquier duda al respecto.
Alejándonos de las especulaciones, por ahora sabemos que:
- El BatCoV RaTG13(MN996532) y el PangCoV MP789 (MT084071) son los parientes más cercanos del SARS-CoV-2.
- El RaTG13 no se une al ACE-2 del pangolín.
- Ya se han creado CoVs quiméricos antes.
- Hasta el 2020 el estudio de CoVs solo requería un laboratorio de bioseguridad nivel 2.
- El SARS-CoV-2 difiere en más de 6000 nucleótidos con el SL-SHC014-MA15 (un CoV quimérico que adquirió patogenicidad aumentada en ratones y su estudio fue restringido bajo la póliza de ganancia de función).
- Las porciones de genoma que comparte el SARS-CoV-2 con el VIH-1 no son específicas y el reporte inicial del Instituto de Tecnología de Nueva Delhi ya fue retractado.
Especies susceptibles
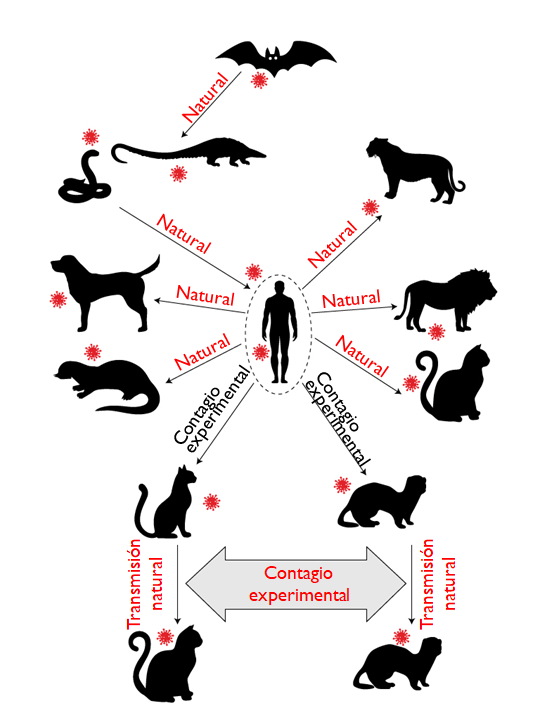
El modelamiento predictivo en base a las secuencias del ACE-2 de otros mamíferos y los estudios previos con el SARS-CoV-1 permitieron proponer de manera transitoria las especies susceptibles a la infección y estas luego fueron confirmadas de manera experimental para detectar reservorios y modelos animales que puedan ayudarnos con su investigación.
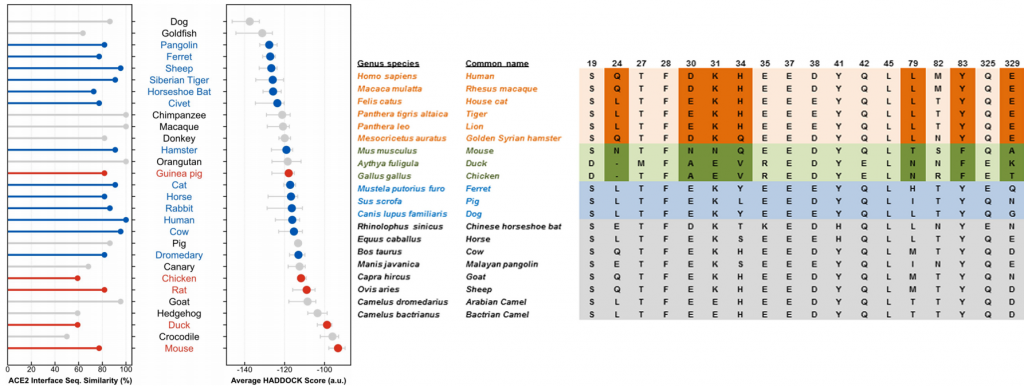
A la fecha, los animales oficialmente susceptibles y que pueden llegar a desarrollar síntomas son los:
- Primates no humanos (Macaca mulatta, Macaca fascicularis, Chlorocebus sabaeus).
- Tigres de malacia (Panthera tigris jacksoni)
- Tigres siberiano (Panthera tigris altaica)
- Liones africanos (Panthera leo)
- Visones americanos (Neovison vison)
- Hurones (Mustela putorius furo)
- Hamsters dorados (Mesocricetus auratus)
- Perros (Canis lupus familiaris)
- Gatos (Felis silvestris catus)
De esta lista, sólo en los visones americanos se ha logrado identificar una variante capaz de hacer un salto de huésped hacia el humano, razón por la que el gobierno danés decidió sacrificar a más de 17 millones de visones en 2000 granjas y dar por terminada su participación en la industria peletera, actividad por la que facturaba alrededor de medio billón de euros anuales.
La probabilidad de los perros y gatos desarrollen la enfermedad es baja y, según los reportes depositados en el Sistema Mundial de Información Sanitaria (WAHIS) de la OIE, la morbilidad no sería severa y su mortalidad estaría asociada más que nada a otras enfermedades debilitantes. En el caso de los perros no parece existir la posibilidad de contagio entre especímenes de su misma especie u otros animales, caso distinto al de los gatos y hurones en los que sí es posible que contagien a otros animales de su misma especie, aunque ya se ha hipotetizado su compromiso en la propagación del virus en las granjas de visones danesas.
Más adelante hablaremos de otros CoVs de interés veterinario, su patogénesis y el estado del arte en su tratamiento.
Haznos llegar tu opinión a través de los comentarios o escribirnos al siguiente correo.
Si te gustó este articulo suscríbete y compártelo en tus redes.
También podría interesarte : ¿Por qué la profesión veterinaria es imprescindible para la sociedad actual?
Referencias
- Borkovich D., Noah P. 2014. Big Data in the Information Age: Exploring the Intellectual Foundation of Communication Theory. Information Systems Education Journal (ISEDJ) 12 (1), 15-26.
- Borkovich, D. J. (2012). When corporations collide: Information overload. Issues in Information Systems, 13(2), 269-284.
- https://www.nejm.org/doi/pdf/10.1056/NEJMoa2001017?articleTools=true
- Phan, T. (2020). Novel coronavirus: From discovery to clinical diagnostics. Infection, Genetics and Evolution, 104211.
- Zhou P, Yang X-L, Wang XG, Hu B, Zhang L, Zhang W, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. (2020) 579:270–3. doi: 10.1038/s41586-020-2012-7
- Tang X, Wu C, Li X, Song Y, Yao X, Wu X, et al. On the origin and continuing evolution of SARS-CoV-2. Natl Sci Rev. (2020) 7:1012–23. doi: 10.1093/nsr/nwaa036
- Zhang, T., Wu, Q., & Zhang, Z. (2020). Probable Pangolin Origin of SARS-CoV-2 Associated with the COVID-19 Outbreak. Current Biology.
- https://www.cdc.gov/sars/guidance/f-lab/app5.html
- Rodrigues, J. P. G. L. M., Barrera-Vilarmau, S., M C Teixeira, J., Sorokina, M., Seckel, E., Kastritis, P. L., & Levitt, M. (2020). Insights on cross-species transmission of SARS-CoV-2 from structural modeling. PLoS Computational Biology, 16(12), e1008449.
- Kuo, L., Godeke, G. J., Raamsman, M. J. B., Masters, P. S., & Rottier, P. J. M. (2000). Retargeting of coronavirus by substitution of the spike glycoprotein ectodomain: Crossing the host cell species barrier. J. Virol., 74, 1393‐1406.
- Maier, H. J., Bickerton, E., & Britton, P. (2015). Coronaviruses – Methods and protocols. London: Humana Press.
- Becker, M. M., Graham, R. L., Donaldson, E. F., Rockx, B., Sims, A. C., Sheahan, T., … Denison, M. R. (2008). Synthetic recombinant bat SARS‐like coronavirus is infectious in cultured cells and in mice. PNAS, 105, 19944‐19949.
- Hu, B., Zeng, L. P., Yang, X. Lou, Ge, X. Y., Zhang, W., Li, B., … Shi, Z. L. (2017). Discovery of a rich gene pool of bat SARS‐related coronaviruses provides new insights into the origin of SARS coronavirus. PLoS Pathog., 13, 1‐27.
- CsiszarA, JakabF, ValencakTG, LanszkiZ, TóthGE, KemenesiG, TarantiniS, Fazekas-PongorV, UngvariZ. 2020. Companion animals likely do not spread COVID-19 but may get infected themselves. Geroscience. 42(5):1229–1236.
- EnserinkM. 2020. Coronavirus rips through Dutch mink farms, triggering culls. Science. 368(6496):1169.





